Badania
Białka wiążące RNA, które uczestniczą w regulacji ekspresji genów przez małe RNA u bakterii
Tematem zainteresowań naszego zespołu są białka, które uczestniczą w regulacji ekspresji genów przez małe regulatorowe RNA bakterii. Celem naszych badań jest wyjaśnienie w jaki sposób regulatorowe RNA są rozpoznawane przez wyspecjalizowane w tym celu białka, oraz jakie jest znaczenie biologiczne tych oddziaływań. Wyjaśnienie tych zagadnień jest ważne dla zrozumienia mechanizmów regulacji procesów życiowych komórki bakteryjnej.
Małe RNA bakterii, określane też nazwą sRNA (ang. small RNA), uczestniczą w adaptacji bakterii do zmian w środowisku, w regulacji homeostazy wewnątrzkomórkowej, a także w regulacji procesów związanych z infekcją organizmu gospodarza. Cząsteczki sRNA mają zwykle około 100 nukleotydów długości i kontrolują translację poprzez wiązanie się z komplementarnymi sekwencjami w obrębie cząsteczek mRNA. Działanie regulatorowe cząsteczek sRNA zależy od wiążących je białek, które kontrolują stabilność sRNA, rearanżują ich strukturę, albo ułatwiają ich oddziaływania z cząsteczkami mRNA. U bakterii Gram-ujemnych do takich białek należą m.in. białko opiekuńcze Hfq oraz białka z domeną FinO (Rysunek 1), a u bakterii Gram-dodatnich białka z domeną KH.
Białko opiekuńcze Hfq jest zbudowane w postaci pierścienia złożonego z sześciu takich samych podjednostek. Pierścień Hfq posiada trzy odrębne miejsca wiązania RNA, które pozwalają na jednoczesne wiązanie różnych RNA. Zależnie od sposobu wiązania RNA Hfq może chronić RNA przed degradacją albo przyspieszać ją. Hfq może też pełnić rolę białka opiekuńczego, które rozwija strukturę RNA oraz ułatwia tworzenie oddziaływań pomiędzy komplementarnymi regionami dwóch cząsteczek RNA. W naszych dotychczasowych badaniach wyjaśniliśmy jakie cechy cząsteczek sRNA sprawiają, że mogą one wiązać białko Hfq w warunkach konkurencji z innymi sRNA [1, 2], a także w jaki sposób białko Hfq ułatwia oddziaływania pomiędzy cząsteczkami sRNA a cząsteczkami mRNA [3, 4], oraz pomiędzy sRNA a anty-sRNA [5].
Białka z domeną FinO należą do licznej rodziny białek występujących tylko u tych bakterii Gram-ujemnych, które posiadają także białko Hfq [6]. Białka te są zbudowane z domeny homologicznej do domeny plazmidowego białka FinO, które jest pierwszym odkrytym białkiem tej rodziny. Oprócz domeny FinO białka te mogą zawierać także dodatkowe domeny na N- lub C-końcu cząsteczki. Domena FinO zawiera miejsce wiążące strukturę spinki terminatora transkrypcji, która znajduje się na 3ʹ końcach cząsteczek RNA. W naszych dotychczasowych badaniach opisaliśmy w jaki sposób domena FinO białka ProQ z E. coli rozpoznaje cząsteczki RNA, oraz zaproponowaliśmy jakie cechy RNA zapobiegają ich wiązaniu przez białko Hfq [7].
Znacznie mniej wiadomo o białkach wiążących regulatorowe RNA u bakterii Gram-dodatnich. Ostatnio zaproponowano, że taką rolę mogą pełnić białka z domeną KH, zwane KhpA i KhpB, które wspólnie występują u wielu gatunków bakterii Gram-dodatnich. Białka te wiążą liczne cząsteczki RNA oraz uczestniczą w regulacji podziału komórki bakteryjnej. Jednakże dokładny sposób ich działania w regulacji ekspresji genów przez sRNA nie jest jeszcze poznany [8].
A
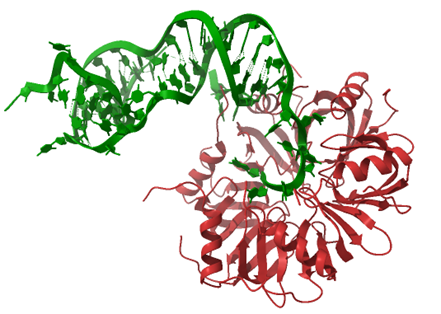
B

Rysunek 1. Oddziaływania cząsteczek RNA z białkiem Hfq (A) oraz z domeną FinO białka RocC (B). (A) Struktura kompleksu sRNA RydC (kolor zielony) z białkiem Hfq (kolor czerwony) z bakterii Salmonella (PDB: 4V2S). (B) Struktura kompleksu sRNA RocR (kolor zielony) z domeną FinO białka RocC (kolor pomarańczowy) z bakterii Legionella (PDB: 7RGU).
Referencje
- Olejniczak M. „Despite similar binding to the Hfq protein regulatory RNAs widely differ in their competition performance.” Biochemistry 50, 4427-4440 (2011)
- Małecka E.M., Stróżecka J., Sobańska D. and Olejniczak M. „Structure of bacterial regulatory RNAs determines their performance in competition for the chaperone protein Hfq.” Biochemistry 54, 1157-70 (2015)
- Wróblewska Z, Olejniczak M. „Hfq assists small RNAs in binding to the coding sequence of ompD mRNA and in rearranging its structure”, RNA (2016), 22(7):979-94
- Kwiatkowska J, Wróblewska Z, Johnson K.A., Olejniczak M. “The binding of Class II sRNA MgrR to two different sites on matchmaker protein Hfq enables efficient competition for Hfq and annealing to regulated mRNAs”. RNA 24(12), 1761-1784 (2018)
- Małecka E., Sobańska D., Olejniczak M. “Bacterial chaperone protein Hfq facilitates the annealing of sponge RNAs to small regulatory RNAs”. Journal of Molecular Biology 433(23), 167291 (2021)
- Olejniczak M., Storz G. “ProQ/FinO-domain proteins: another ubiquitous family of RNA matchmakers?” Molecular Microbiology 104(6), 905-915 (2017)
- Stein E.M., Kwiatkowska J., Basczok M.M., Gravel C.M., Berry K.E., Olejniczak M. Determinants of RNA recognition by the FinO domain of the Escherichia coli ProQ protein. Nucleic Acids Research 48 (13), 7502-7519 (2020)
- Olejniczak M., Jiang X, Basczok MM, Storz G. KH domain proteins: Another family of bacterial RNA matchmakers? Molecular Microbiology 117(1):10-19 (2022)
